「常識覆す仕組み」 大腸菌の大規模ゲノム組み換え解明 東京大など
大腸菌のDNA組み換え仕組みの解明が発表された。大腸菌は特定の酵素とRNAを使い、必要な部分だけを狙った場所に組み換えを行っている。
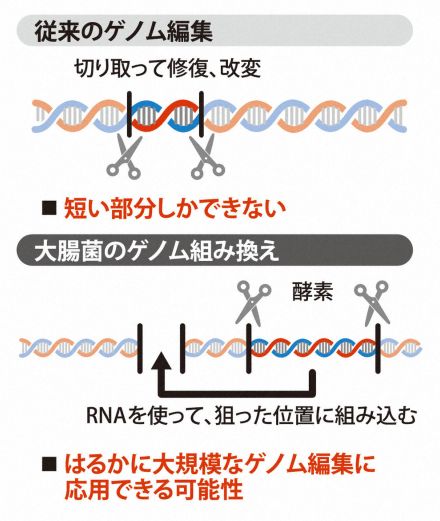
現在広く使われているクリスパー・キャス9とは異なる手法で、DNAの精度よく欠損や改変が可能。将来的には、1万塩基対ほどのDNAの組み込みも可能になる可能性がある。
研究チームはこの発見が次世代のゲノム編集技術につながる可能性を示唆しており、他の生物でも効率的な反応が期待されている。
大腸菌の中で大規模にDNAが組み換わる仕組みを解明したと、東京大と米アーク研究所のチームが発表した。この仕組みを応用すれば、生命の設計図とされるゲノムを桁違いの規模で改変・生成できる次世代のゲノム編集技術につながる可能性がある。
DNAは多くの生き物では不変だが、大腸菌は、自らのDNAの一部分を切り取って別の場所に組み込む「組み換え」をすることが知られている。だが、どんな仕組みで起きるかはよくわかっていなかった。
チームが電子顕微鏡などで詳しく解析すると、組み換えに関わる部分から、組み換えを促進する酵素「リコンビナーゼ」と、組み換える場所を特定する「ブリッジRNA」が作られていた。
大腸菌はこの酵素とRNAを機能させることで、組み換えをランダムに行っているのではなく、必要な部分だけを狙った場所に正確に行っていることがわかった。
現在のゲノム編集技術は、2020年にノーベル化学賞の対象となった「クリスパー・キャス9」という手法が広く使われている。今回とは別の酵素とRNAを使い、狙ったDNAを精度よく欠損させたり改変したりできる。一方、組み込めるDNAが100~200塩基対と少ないのが課題だ。
今回の手法が他の生き物にも使えるかはまだわかっていないが、もし応用できれば、1万塩基対ほどのDNAでも組み込むことが可能だという。クリスパー・キャス9と比べ、ゲノムの情報量を桁違いに書き換えられ、ゲノムを人工的に作り出す合成生物学の手法にも近いという。
チームの西増弘志・東京大教授(構造生物学)は「非常に複雑な反応を可能にする、これまでの常識を覆す仕組みだ。大腸菌以外の生物でも効率よく反応させることができれば、次世代のゲノム編集ツールになり得るだろう」と話した。
成果は27日、英科学誌ネイチャー電子版に2本の論文(https://doi.org/10.1038/s41586-024-07552-4)(https://doi.org/10.1038/s41586-024-07570-2)が掲載された。【渡辺諒】